A cromatografia de troca iônica é uma das técnicas de FPLC mais usadas para separação e purificação de proteínas. Esta aplicação descreve uma separação muito simples de duas proteínas diferentes e explica como a cromatografia por troca iônica funciona.
pI (Ponto Isométrico) e pH
A cromatografia de troca iônica (IEC) separa moléculas de acordo com o tipo e força de sua carga elétrica. O ponto isoelétrico (pI) é definido pelo pH em que a proteína ou molécula não tem nenhuma carga elétrica. A carga superficial da proteína depende do pH da solução na qual ela está eluída.
Quando a solução possui um pH acima do seu pI, as proteínas tem uma carga negativa e se ligam a resinas aniônicas carregadas positivamente. Já no caso do pH da solução estar abaixo do seu pI, as proteínas carregam carga positiva e se ligam a resinas catiônicas carregadas negativamente.
Esta interação elétrica através do pH é usada para a separação e purificação de diversas proteínas. Através do uso de um pH conveniente e baixa concentração de sal, as proteínas se ligam à resina da coluna no passo inicial (Figura 1A). Em seguida, de forma geral, as proteínas são desconectadas da resina da coluna quando submetidas à um gradiente linear de uma solução salina. Os íons do sal competem com as proteínas pelos sítios ativos da resina.
Proteínas com interações iônicas fracas são as primeiras a eluir da coluna. No caso de cromatografia de troca catiônica, as proteínas com cargas negativas mais fracas são as primeiras a eluir (Figura 1B). Com um aumento na concentração do sal, as proteínas com interações mais fortes eluem da coluna (Figura 1C).
Resultados da troca iônica
α-Chimotripsinogeno A (pI 8,97) e Lisosima (pI 11,35) são proteínas que têm alto pI, o que as torna candidatas ideais para a cromatografia de troca catiônica (Figura 2). Ambas proteínas se ligam sob baixa concentração salina à resina. Durante o passo de lavagem, que remove proteínas não ligantes potenciais, só uma pequena quantidade de impurezas eluíram da coluna. α-Chimotripsinogeno A eluiu primeiro da coluna devido ao seu pI mais baixo de 8,97 (Figura 2, pico 1).
Com um gradiente incremental e, portanto, um aumento incremental na concentração salina, a Lisosima eluiu como mostra o segundo pico (Figura 2, pico 2). As proteínas eluídas foram coletadas de forma automática no coletor de frações. O gradiente de sal foi monitorado por um condutivímetro (Figura 2, sinal vermelho).
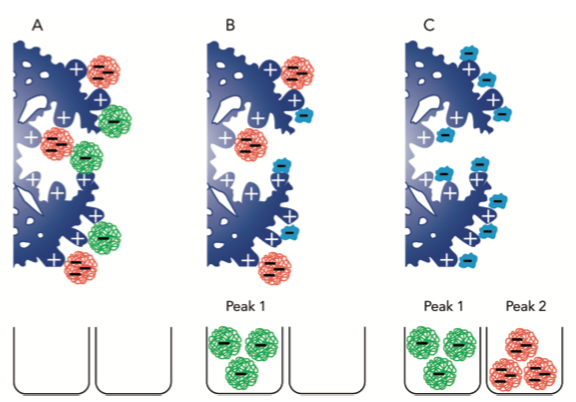
Figura 1:
Princípio de separação catiônica
A) proteínas com cargas negativas diferentes se ligam a resina de troca catiônica,
B) através do incremento da concentração de sal, proteínas com carga negativa mais fraca eluem primeiro,
C) quando a concentração de sal aumenta, as proteínas com cargas negativas mais fortes eluem por último.
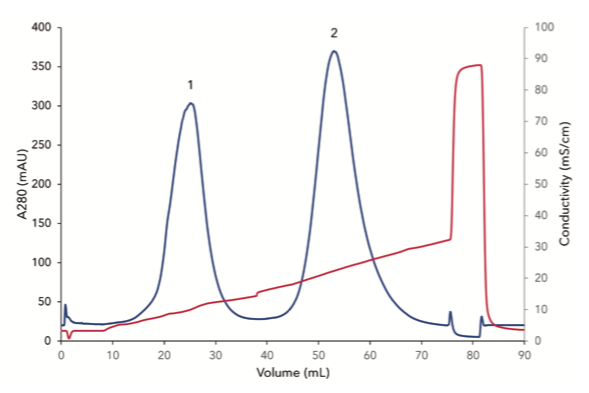
Figura 2:
Cromatograma de uma separação de dois tipos de proteínas com cromatografia de troca iônica, linha azul – sinal UV 280nm, linha vermelha – sinal do condutivímetro,
1) pico contendo α-Chimotripsinogeno A (2,5 mg/mL),
2) pico contendo Lisosima (2,5mg/mL)
Materiais e Métodos
Nesta aplicação, foi utilizado um sistema de purificação AZURA® Bio composto de:
- Uma bomba AZURA® P 6.1L LPG livre de metais com um cabeçote de 10mL;
- Um assistente AZURA® ASM 2.1L com bomba de alimentação e duas válvulas de injeção;
- Um detector de arranjo de diodos AZURA® DAD 2.1L com 10mm, célula de fluxo de 10µL;
- Um monitor de condutividade AZURA® CM 2.1S;
- Um coletor de frações Foxy R1.
- Antes da corrida, a coluna de troca catiônica de 1mL (HiTrap Capto S) foi equilibrada em 10mL do tampão A (tampão fosfato 20 mM, pH 7.1). A vazão para a corrida foi 2mL/min.
- A mistura de proteína de 500µL (α-Chimotripsinogeno A 2,5mg/mL + Lisosima 2,5mg/mL) foi injetada.
- Em seguida a coluna foi lavada com 4mL de tampão A para remover toda a proteína não ligante.
- As duas proteínas foram eluídas com um gradiente linear ao longo de um volume total de 70 mL. A concentração de sal aumentou com o aumento da concentração do tampão B (tampão fosfato 20mM, pH 7.1 + 1M NaCl) no gradiente de 0% a 30%.
- As proteínas que eluíram foram coletadas pelo coletor de frações.
- A coluna foi regenerada com uma lavagem de alta concentração de sal com 6mL de tampão B, a seguir um reequilíbrio da coluna com 10mL do tampão A.
- As proteínas foram detectadas a 280nm e o sinal de condutividade foi registrado para monitorar o gradiente de sal.
Conclusão
Esta aplicação bem direta ilustra o princípio da cromatografia de troca iônica. Dois tipos de proteínas com diferentes cargas superficiais eluíram da coluna de troca catiônica quando submetidas à uma concentração crescente de sal. As proteínas puderam ser facilmente separadas pela cromatografia de troca catiônica com um sistema de purificação AZURA® Bio.
Materiais Adicionais e Parâmetros
Tabela A1: Parâmetros do método
| Eluente A | 20 mM tampão fosfato pH 7.1 |
| Eluente B | 20 mM tampão fosfato pH 7.1 + 1 M NaCl |
| Gradiente | Volume [mL] % A % B 0 100 0 4 100 0 74 70 30 74.02 0 100 80 0 100 80 100 0 90 100 0 |
| Vazão | 2 mL/min |
| Tempo corrida | 45 min |
| Temperatura da coluna | 25°C |
| Modo de injeção | Full loop |
| Volume de injeção | 0,5 mL |
| Data rate | 2 Hz |
| Comprimento de onda | 280 nm |
| Constante de tempo | 500 ms |
Tabela A2: Configuração do sistema e informações
| Instrumento | Descrição |
| Bomba | AZURA P 6.1L LPG, livre de metal |
| Detector | AZURA DAD 2.1L |
| Célula de fluxo | 10mm, 10µL, Ti, 300bar |
| Assistente | AZURA ASM 2.1L Esquerda: Bomba com sensor de pressão, 50 mL cabeçote, titânioMeio: válvula de injeção de 6 portas/ 2 posições, conectores 1/16″Direita: válvula de injeção de 6 portas/ 2 posições, conectores 1/16″ |
| Coletor de frações | Foxy R1 |
| Condutivímetro | AZURA CM 2.1S |
| Coluna | Capto S, GE Healthcare, HiTrap, 1mL |
| Software | PurityChrom 3D Option |
Baixe aqui a aplicação completa.